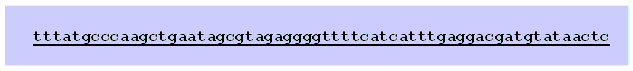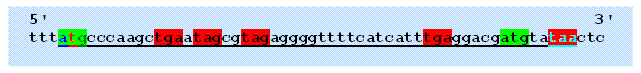Un genoma completamente secuenciado: ”¿Qué tantos triques podemos encontrar en un genoma?”
Por Verónica Jiménez Jacinto,
Gabriel Moreno-Hagelsieb e
Irma Lozada-Chávez
Revisión en divulgación: M. en C. Alma Mendoza Ponce
Revisión en Fidelidad Científica: Dr. Gabriel Moreno-Hagelsieb
“- La cacariza... - El seis dos... - Pues yo me doblo - Eeessoossss que se doblan a la primera...”
Con este diálogo entre cuatro tahúres del dominó se inicia cada una de las TRES HISTORIAS que se narran en la película “El Callejón de los milagros”, el diálogo alardeaba sobre algo aparentemente tan sencillo como una partida de dominó, donde pueden irse entretejiendo las jugadas hasta convertirse en un complicado laberinto que, pese a los espectadores, siempre encuentra una salida (en la película, la salida de cada personaje no siempre es la mejor... o mejor dicho, casi siempre es la peor).
En el caso de la secuenciación de un genoma ocurre algo similar, claro que sólo si imaginamos que cada segmento de nucleótidos (A, T, G, C) que conforman un genoma es como una sección de la pieza de dominó. El juego inicia con la sopa sobre la mesa –no precisamente la de fideos–, para aquellos quienes no gastan sus quincenas, o cantidades sorprendentes de corcholatas o el orgullo en el dominó, sólo tienen que saber que "la sopa" es el simple revoltijo con las manos de todas las fichas con las que se lleva a cabo el juego. Descifrar la secuencia completa de un genoma se asemeja a la sopa del dominó, pues si tenemos todas las piezas del juego pero no sabemos el orden con el que quedarán al final. Entonces, con un dominó clásico formado de 28 piezas logramos armar 320, 000 combinaciones o juegos distintos. Sin embargo, con 4 piezas o nucleótidos (A, T, G, C) de los que se compone el DNA de cualquier genoma, por ejemplo el de la bacteria Rhizobium etli con una longitud de 6, 530, 234 pares de bases (seis millones quinientos treinta mil doscientos treinta y cuatro pares de nucleótidos), el número de combinaciones que se pueden armar da una cifra de más de 4 millones de dígitos ¿Puedes siquiera imaginar un número de estas dimensiones?
El pasado 9 de marzo del año 2006 se publicó, en más de una docena de periódicos a nivel nacional, una noticia que no es común en este país, primero porque era una noticia agradable, segundo porque era del ámbito científico y tercero porque involucraba cifras numéricas de las que hemos estado hablando: "Un grupo de mexicanos, entre biólogos y computólogos se hicieron a la tarea en ser los primeros en secuenciar y anotar el genoma completo de la bacteria fijadora de nitrógeno Rhizobium etli, lo cual ha posicionado a México como el segundo país Iberoamericano en alcanzar esta meta".
¿Cómo se logró esto? El desciframiento de un genoma...
Una vez terminada la sopa, al menos siete fichas de dominó se han repartido para cada jugador –¿quién tiene la mula de seis?– Quien tiene la ventaja de poseer la ficha que regirá el resto del juego (6:6) inicia la partida con una sonrisa. Los jugadores en lo sucesivo tienen que hacer coincidir el número de puntos del extremo final de una ficha puesta en combinación con el extremo inicial de la ficha que le sigue en turno. Por ejemplo, después de que un jugador colocó la mula de seis, el siguiente en turno puede colocar cualquiera de las fichas que tengan seis puntos en uno de sus extremos y cualquier número en el otro extremo (6:? o ?:6), por ejemplo la ficha 6:2; el siguiente jugador en turno hará lo mismo tratando ahora de hacer coincidir fichas con extremos de seis o dos puntos (6:? o ?:6; 2:? o ?:2) y así sucesivamente. El jugador más buzo –o el que ha vendido su alma al diablo– ha hecho coincidir todas sus fichas en el juego, lo cual lo convierte en el ganador.
Aunque jugar dominó no le parezca tan complicado al lector, el “desciframiento de un genoma completo” sigue la lógica de un juego de dominó: hacer coincidir piezas, descifrar códigos, y encontrar los métodos que nos permitan terminar en el menor tiempo posible, pero sin perder precisión ni calidad en el resultado final. Esta lógica se enfoca en la solución de tres historias diferentes, pero muy entrelazadas, que nos causan reales dolores de cabeza para quienes realizamos esta labor: 1) ensamblar los segmentos de DNA que conforman el genoma, 2) encontrar y anotar los genes, y 3) publicar el genoma completamente secuenciado en las bases de datos biológicas de corte internacional.
HISTORIA 1. El ensamblaje de un genoma completo:
“Como un juego de dominó”
Sabemos que el genoma de un organismo está constituido por una larguísima cadena de DNA, y es esta peculiaridad la que impide que pueda procesarse y tratarse la cadena completa en los laboratorios, por lo que en realidad se corta el DNA en segmentos para que se pueda obtener su secuencia de nucleótidos. Estos segmentos pueden ir desde 1000 hasta 10000 pares de bases (cada par de nucleótidos es llamado un “par base”, pb), dependiendo de la técnica de secuenciación y del organismo a secuenciar. Una vez que se obtienen las miles de secuencias de nucleótidos en el laboratorio, todos estos segmentos tienen que ser ensamblados para obtener nuevamente la serie específica de los nucleótidos que conforman el genoma (DNA) de un organismo. Esto es justo lo que se conoce como el proceso de ensamblado de un genoma. Si en lugar de tener las 28 fichas del juego de dominó convencional, tuviéramos algunos cuantos miles de fichas; y que en lugar de tener de cero a seis puntos por extremo en cada ficha, tuviéramos una cadena de letras de longitud variada, desde cien hasta mil o dos mil caracteres de nucleótidos (ATGC). Más aún, los investigadores que participamos en el ensamblado de estas secuencias sólo podemos considerarnos ganadores cuando todas las fichas disponibles en el juego se han acabado; ya que el objetivo final de la secuenciación es obtener un solo genoma, por lo que en teoría no pueden sobrar fichas –aaahhh, pero tampoco se vale decir ‘safo’, ‘paso’, ‘me doblo’ ni alguna de sus variantes –.
Luego hay que armar una especie de rompecabezas con todos esos miles de pequeños segmentos, ¿pero si no sabemos cuál es la secuencia específica del genoma completo, cómo sabremos qué segmento le sigue a otro? Las personas dedicadas a la sastrería experimentan un problema similar. Si le preguntamos a cualquier sastre qué es lo más difícil al confeccionar una prenda de vestir, casi todos harán consenso con que no es el trazo, ni el corte, tampoco la costura, sino el ensamblado de la prenda. ¿Cómo saben los sastres qué segmentos de tela van con cuáles otros? Los bordes donde se realizan las costuras son la clave. La costura de todos los pedazos de tela de una prenda se lleva a cabo por esas pequeñas pestañas que poseen a lo largo de cada extremo. El borde de cada pedazo de tela debe coincidir o empalmar de la manera más precisa con el otro pedazo –por supuesto antes de coserlos con la máquina, ¡ja!–.
Uno de los métodos más usados para realizar el ensamblado de las secuencias de un genoma es a través de la complementaridad de las cadenas DNA que lo componen. Es necesario que recordemos que el genoma es en realidad es una doble cadena de DNA, y los nucleótidos que a su vez conforman el DNA son complementarios, es decir, hacen parejas precisas entre ellos: A (adenina) siempre interactúa con T (timina), mientras que C (citosina) siempre interactúa con G (guanina). Entonces, si a cada segmento de DNA le dejamos unos cuantos nucleótidos (como los bordes de la tela) podríamos buscar el segmento de DNA entre todos los miles de segmentos que tenemos que son complementarios a nuestro segmento de DNA. Este pequeño segmento de DNA en todos los segmentos que son secuenciados en el laboratorio previamente hará la función del borde de costura que nos permitirá saber cuál es el segmento de DNA que le sigue en la secuencia del genoma, como podemos observar en las figuras número 2 y 3.

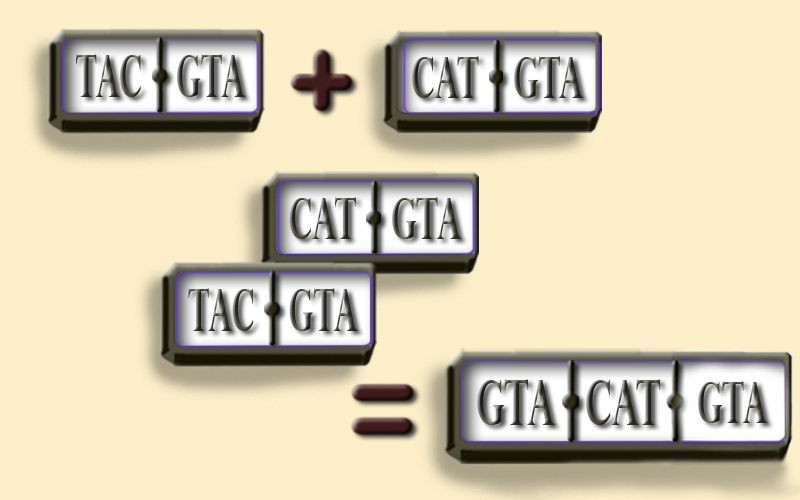
Cuando tenemos un rompecabezas de inmensas dimensiones –vamos… que ni siquiera cabe en la mesa familiar de nuestra casa– y tenemos que terminarlo en esta vida, generamos un método; por ejemplo, reunir todas las piezas que estén relacionadas con una imagen que forma parte del rompecabezas. Este método nos permitirá terminar el rompecabezas en el menor tiempo, pero ello no debe impedir que sea eficaz y precisa la imagen que se debe obtener al final. De la misma forma, los bioinformáticos (dícese de los investigadores que trabajan con problemas biológicos con ayuda de las computadoras) llevan a cabo procesos de ensamblaje y procesamiento de grandes cantidades de información usando algoritmos, es decir un conjunto de pasos finitos y definidos de manera precisa que nos permiten tomar en cuenta todas posibilidades de un problema, en caso de que nos sea necesario tomar diferentes decisiones para resolver ese determinado problema. Estos algoritmos son implementados en códigos que puedan ser procesados en computadoras, como por ejemplo, los lenguajes de programación.
La implementación computacional de la ensamblación y de la anotación (que veremos más adelante) de los genomas nos permiten a los dedicados a esta labor, por un lado, dar solución a los problemas de ensamblado, análisis, anotación y evaluación de las secuencias obtenidas en un genoma con mucha certeza y precisión y también llevar a cabo esta actividad con una menor cantidad de esfuerzo, espacio y tiempo. Mientras que de manera manual estas actividades podrían llevarnos desde varios meses hasta años, de manera computacional a través del uso de algoritmos, el trabajo puede tomarnos apenas unas semanas.
HISTORIA 2. La anotación de un genoma:
“Ahora sí que al derecho y al revés...”
Una vez que se ha terminado exitosamente el ensamblaje de un genoma, aunque la definición más conocida de “genoma” es “toditito el material genético de un organismo”, con lo que los bioinformáticos terminamos es con una o varias palabras, bueno palabrototototas. Por ejemplo, en el caso del genoma humano nos quedamos con 24 tamañas palabrotas puesto que está compuesto de 24 cromosomas el genoma humano. O bien, en el caso del genoma de la bacteria Rizhobium etli que tiene 1 sola palabrota (genoma), acompañada de otras 6 palabrotas más pequeñas y accesorias al genoma de esta bacteria (llamadas plásmidos). Y entonces, para aquellos lectores que crean acérrimamente que lo peor que le puede pasar a uno es no saber quién va a ganar el campeonato mundial de fútbol o cómo ser “totalmente Palacio” sin vaciar las tarjetas de crédito propias y ajenas en el Palacio de Hierro, ¡pues sepan que se quedan cortos! Justo es en esta fase donde los investigadores padecemos de las peores pesadillas, porque es ahora donde abundan muchas de las preguntas existenciales de las Ciencias Genómicas, como ¿qué hay en el genoma y cómo podemos averiguarlo? O bien ¿qué son los genes? Que si ¿los genes tienen una función o varias? O también ¿cómo podemos conocer la función que tiene un gen? Que ¿cómo le hacemos para encontrar el gen en una secuencia larguísima de nucleótidos? Que si ¿tienen alguna función las regiones del genoma que no poseen genes?, entre muchas otras…

Una de las razones extremas de angustia para nosotros los investigadores es si alguno de los genes que se encuentran en el genoma humano puede estar relacionado o está relacionado a alguna enfermedad (anemia drepanocítica y la diabetes por ejemplo), y es posible diseñar una terapia genética que contrarreste la enfermedad. Entonces es totalmente necesario primero identificar ese gen entre el montón de las 24 palabrotas (cromosomas) que conforman el genoma. Además, es indispensable saber si la enfermedad es producto de un cambio (mutación) en la secuencia de este gen. Entonces tenemos que identificar cambio(s) en la secuencia. Si ese cambio o cambios en la secuencia del gen son muy precisos en la población humana, diseñar el medicamento equivocado no sólo puede ser fatal para quien padece ese mal, sino que podría afectar a los que no padecen la enfermedad en caso de que ellos tomen el medicamento por un diagnóstico equivocado. Por fortuna, la naturaleza expone ciertos patrones que podemos identificar y usar para contestar completa o parcialmente estos cuestionamientos. Específicamente para el caso de los genomas, estos patrones los hemos identificado como tipos de códigos que hay que descifrar en las secuencias de un genoma.
El código genético: "Uno de los acertijos más interesantes de la naturaleza"
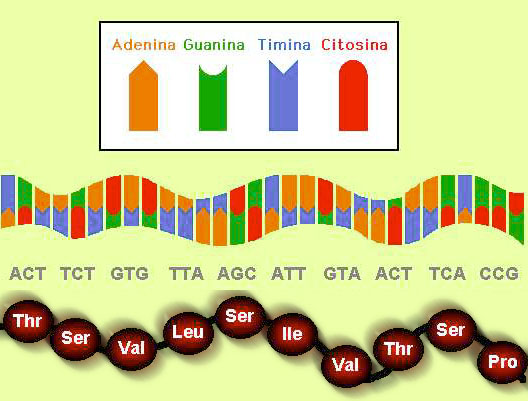
Si el lector alguna vez ha tenido la frustración de no poder recitar un trabalenguas completo o quedarse en blanco ante el desciframiento de un acertijo, ¡querido lector este es el momento para reivindicarse! He aquí algunos de los secretos de la “gramática” con la que podemos leer el genoma, uno de los acertijos más importantes de la naturaleza. Para empezar, lo más sencillo es tratar de identificar genes que dan lugar a proteínas, tales como la colágena que forma el cabello y las uñas, o la melanina que da el color a la piel, o la insulina que regula la glucosa en la sangre, entre miles otras. Entonces, el alfabeto del DNA (que contiene a los genes) es de solamente cuatro letras (A –a, T –t, C –c, G –g), pero el alfabeto de las proteínas es, oficialmente, de 20 letras.
Las proteínas tienen un código muy definido, se componen de una combinación de 20 unidades bioquímicas pequeñas llamadas aminoácidos, cada uno de éstos a su vez se compone de tripletes de nucleótidos (tres nucleótidos) que forman cadenas. Entonces, a partir del arreglo de los nucleótidos en tripletes (también llamados codones) es como podemos buscar genes en la larga cadena de DNA que forma el genoma. Sin embargo, aún existen miles de tripletes que por sí mismos no dicen si en realidad ellos corresponden a un gen o si sólo son tripletes formados al azar en el genoma. Afortunadamente, la naturaleza sigue compadeciéndose de nosotros, así que podemos identificar, en estas larguísimas cadenas de DNA, cuatro codones que marcan el inicio y el término de un gen. Sólo uno de los cuatro codones corresponde al codón de inicio (ATG), que además codifica para el aminoácido Metionina (abreviado como M o Met); mientras que con alguno de los tres codones restantes (TAA, TAG o TGA) podemos saber dónde termina un gen en la larga cadena de DNA. Hacer falta saber algo más, tenemos que recordar que el genoma es una composición de una doble cadena complementaria de DNA, entonces el genoma puede leerse ahora sí que tanto al derecho como al revés. Una de las cadenas de DNA tiene una dirección de 5’ a 3’, mientras que la cadena de DNA que va en sentido inverso tiene una dirección 3’ a 5’.
Entonces, si retomamos las tres reglas “gramaticales” con las que podemos empezar a descifrar los códigos de una secuencia larguísima de DNA:
-
Primero es necesario que encontremos los codones (tripletes de nucleótidos) de inicio (ATG –atg) y también los codones de término (TAA –taa, TAG –tag o TGA –tga).
-
Después llevamos a cabo la regla anterior tanto al derecho (5’ a 3’) de una de las cadenas como al revés (3’ a 5’), puesto que en ambas direcciones se encuentran los genes.
-
Los genes están representados por letras compuestas de 3 nucleótidos cada una (codones). En el caso de las proteínas, entonces serán 20 letras que conforman cada una a los 20 aminoácidos. Los genes contienen a varios de esos aminoácidos.
Con sólo las dos primeras reglas podemos decir que existen seis posibles formas de leer una secuencia de DNA dependiendo del punto de inicio y de la dirección en que se lea la cadena (3 en cada dirección). Cada una de estas formas de lectura es lo que conocemos como una pauta de lectura (Reading Frame). Si tomamos al azar una secuencia en la dirección 5’ a 3’ del genoma de algún bicho (entiéndase por bicho como el término coloquial que los biólogos –y no biólogos – usamos para referirnos a cualquier cosa viva: animal, bacteria, planta, hongo, humano, etc.). ¡Ahora es cuando, impertérrito lector!, el siguiente paso es buscar si esta secuencia presenta pautas de lectura aplicando la regla número 2) buscar codones de inicio y de término (ATG –atg, TAA –taa, TAG –tag o TGA –tga):
Después de unos minutos, podemos identificar dos codones ATG (marcados en verde abajo) que nos indican que ahí puede iniciar un gen, así como también encontramos 5 codones TGA (marcados en rojo abajo) que nos indican que ahí podría terminar un gen.
Entonces ¿dónde está el gen? Por lo general, el codón de inicio y el codón de término más distantes entre sí son los que se toman para identificar las pautas de lectura. Ahora sí, sólo tomando en cuenta estos dos condones podemos ver las tres pautas de lectura subdivididas en tripletes o codones:
La pauta de lectura 1 empieza con el nucleótido “a”, mientras que la segunda empieza con “t” y finalmente la tercera pauta de lectura empieza con “g”. Cuando alguna de estas pautas de lectura contiene tripletes que representan aminoácidos (aquí abreviados por sus letras oficiales: por ejemplo, A de alanina, P de prolina, S de serina y R de arginina) y que, por tanto, en conjunto es posible que puedan dar lugar a una proteína, se denomina pauta de lectura abierta (open reading frame: ORF). Desde luego que nuestro lector prefiere comerse los aminoácidos en lugar de leerlos –igual que nosotros–, la fenilalanina, por ejemplo, es un aminoácido que se usa como edulcolorante de chicles, refrescos y otros productos como sustituto del azúcar. Si ésta se encontrara en un ORF el triplete TTT –ttt o bien el TTC –ttc, entonces habríamos identificado el código de la fenilalanina (abreviado como F o Phe). De forma natural, la fenilalanina es uno de los constituyentes presentes en todas las proteínas (vegetales o animales). Dado que el humano no puede sintetizarlo “de novo” este aminoácido es considerado como esencial en la dieta diaria. La fuente más importante de fenilalanina para el humano son los alimentos ricos en proteínas, como la carne, el pescado, los huevos y los productos lácteos. Por supuesto que esas proteínas que comemos están formadas por conjuntos de aminoácidos (como la fenilalanina) y estos aminoácidos a su vez derivaron de genes que se encuentran en alguna parte de la secuencia completa del genoma de esos animales y de esas plantas. Aunque en términos estrictos, la fenilalanina que se consume como edulcolorante es un aminoácido sintético.
Hacia el final de esta segunda historia observamos que las tres pautas de lectura abierta u ORFs dan lugar a aminoácidos, es decir ¿puede cualquiera de estos ORFs ser un gen que da lugar a una proteína? En general la respuesta es NO, aunque cualquiera de estas tres posibilidades puede contener una proteína, sólo uno de los ORFs candidatos es aquel que representa al gen de la secuencia que escogimos al azar del genoma de un bicho cualquiera. El criterio de prioridad para saber cuál de todos los ORFs es el que corresponde al gen nos dice que el ORF más largo que no contenga codones de término en su interior es aquel que corresponde al gen. Bajo esta regla de prioridad es como sabemos que el ORF ganador es:
Un gen no sólo da lugar a proteínas, también da lugar a otros elementos con funciones igual de importantes que las proteínas. Todos estos elementos en su conjunto están relacionados con las funciones morfológicas, fisiológicas, ecológicas, reproductivas, etc. que determinan el buen o mal desempeño de un organismo. Detectar los ORFs en la secuencia de un genoma completo es la primera aproximación para encontrar los genes, a este proceso se le conoce como la anotación de un genoma. Aunque la anotación también se lleva a cabo en secuencias “sueltas” que forman parte de algún bicho cuyo genoma aún no es secuenciado. Terminar la anotación de un genoma es el mejor tónico para recuperar el sueño de quienes trabajamos en esta labor. No sólo porque tenemos estas reglas para descifrar los códigos de los genomas, sino porque además el código genético, con ciertas excepciones, es 1) universal, y 2) redundante. Con universal nos referimos a que si tenemos que anotar la secuencia del genoma de un bicho cualquiera, su genoma tiene exactamente las mismas reglas y el mismo código de codones para formar proteínas. Mientras que el hecho de que sea redundante implica que existe más de un triplete (codón) para un mismo aminoácido, como en el caso de la fenilalanina (TTT –ttt o TTC –ttc). De esta forma, podemos contar con un mayor número de codones que generan la gran diversidad de proteínas que existen en la naturaleza. Invitamos al lector a ver la figura número 4 para una mayor explicación al respecto.
Existen algoritmos muy exitosos para realizar todos los procedimientos que aquí exonemos, pero de forma más eficiente, rápida y confiable, algunos de ellos se identifican como Blast-Glimmer, GeneMark, RBSFinder, tRNAScan. A pesar de que sus nombres sean casi imposibles de pronunciar y carezcan de real significado aquí, la disposición de estos algoritmos (en su mayoría gratis) en todo el mundo, nos permite detectar ORFs y genes en los genomas completamente secuenciados. Además de que a través de ellos nos es posible manipular en forma “relativamente rápida” grandes cantidades de información como resultado de los análisis exhaustivos que deben hacerse con la secuencia de DNA que obtuvimos en la etapa de ensamblamiento del genoma. Cuando hablamos de “grandes cantidades de información”, en realidad se supera la cantidad de palabras que podemos escucharle a la vecina más cercana en los chismes del lavadero; para darnos una humilde noción de las comparaciones, cuando se terminó de ensamblar el genoma de la bacteria fijadora de nitrógeno R. etli se generaron 4 megas, donde un mega está compuesto de 1 024 000 000 bites y en cada bite cabe un solo nucleótido (A o T o C ó G) del DNA. Ahora que si quiere verse desde otra perspectiva, pues casi cualquier canción en formato MP3 tiene las mismas dimensiones de información que el genoma de esta bacteria.
HISTORIA 3. La publicación de los genomas completamente secuenciados:
“El que no enseña, no vende”
Saber cómo debemos leer el genoma del bicho que hemos secuenciado no sólo implica conocer el número de genes que posee, sino también muchas características específicas de cada gen, como por ejemplo su tamaño, su posición específica en el genoma, la función que desempeña, si se encuentra “solo” o quiénes son sus genes vecinos. Por ello, toda la información que obtenemos en los procesos anteriores se va depositando en una enorme base de datos de corte biológico desarrollada por el grupo de trabajo responsable del genoma secuenciado, que permitirá más adelante a quien lo desee consultar de manera rápida, sencilla, confiable y estructurada cualquier información relacionada al genoma que se secuenció. Las secuencias generadas de proyectos de secuenciación implican la materia prima de las Ciencias Genómicas en el más amplio sentido de la palabra, ya que esta fuente de información nos permite también generar nueva información a partir del análisis de estas secuencias respecto a temas específicos de su función, su localización, su obtención, sus aplicaciones y diversos otros.
En los comienzos de las Ciencias Genómicas, conforme se obtenían secuencias completas o parciales de un genoma, se recurría inevitablemente a la comprobación experimental para saber si los ORFs que se predijeron como genes eran correctos. Con el paso de los años, los bancos de datos de secuencias fueron almacenando toda esta información, así que actualmente para la mayoría de los recién desempolvados genes, ya no es necesario llegar hasta la comprobación experimental, puesto que basta con hacer comparaciones automatizadas con algoritmos de las secuencias nuevas contra las que se encuentran en las bases de datos genómicas. Con ciertos grados de confianza podemos decir qué proteína es codificada por el gen que se identificó como ORF.
Aún cuando estos dos grandes procesos, la ensamblación y la anotación no han concluido del todo, inicia una tercera tarea igual o incluso más apremiante: “ganar el registro en el GenBank consorsium” el cual es una organización a nivel internacional que se encarga de “controlar” el registro de los nuevos genomas, los cambios en la anotación de los mismos, así como de la actualización sobre la información relacionada a los genes de estos mismos genomas. Los consorcios de Bases de Datos Biológicos, tales como el GenBank, constituyen un consolidado gremio de la comunidad científica que, a través de un completo formato de registro de los nuevos genomas completamente secuenciados, comprueban y validan el proceso de ensamblaje y la anotación del genoma propuesto para que sean publicados, y que de esta forma queden disponibles al público, con el respaldo de la comunidad científica. Ver figura 5.

Hemos llegado al final de nuestra tercera historia. Varios años de trabajo de este proyecto fueron acompañados, fuera de las horas de trabajo, de un considerable número de partidas de dominó, acertijos, tazas de café, y de vez en cuando de una que otra película que nos permitiera a los investigadores continuar con la mente despejada el desciframiento del genoma completo de la bacteria Rizhobium etli. Así como en película de “El callejón de los milagros”, nos permitió ver como cada personaje “jugó su propia partida de domino” para terminar con una secuencia de hechos distinta llamada “vida”, los científicos hemos logrado descifrar los códigos genéticos básicos de la bacteria Rizhobium etli que determinan en sí la materia prima de la que se compone toda la vida de esta bacteria. ¿Qué haría nuestro lector con una vida en sus manos? Aparte de la propia claro…